纪念斯隆·凯特琳癌症中心(MSKCC)和GRAIL联合研究团队,今天带来一项重要研究成果。
这项由MSKCC实验病理学主任JorgeReis-Filho和临床医学科学家Pedram Razavi共同领导的研究,对124例癌症患者的cfDNA、肿瘤组织样本和白细胞DNA,以及47名无癌志愿者的cfDNA和白细胞DNA,进行了超高深度测序(60000×)。
他们发现,肿瘤患者血液cfDNA中携带的大部分基因突变,可能不是来自于癌细胞,而是血液中白细胞。
具体说来,在癌症患者的cfDNA中,克隆性造血带来的变异可能占53.2%;而在健康的志愿者体cfDNA中,这一数字能达到81.6%。这意味着,克隆造血带来的突变比科学家原来认为的要常见的多。
“看到这个结果,我们差点儿从椅子上掉下去。”Reis-Filho说[1]。
这也给液体活检相关的研究提出了挑战,在给患者开展液体活检时,白细胞DNA应该是必检项目,甚至需要开展超高深度的测序。
Pedram Razavi、DavidN. Brown和华人科学家Bob T. Li是共同第一作者。研究论文发表在顶级期刊《自然·医学》上[2]。

▲ 从左到右:Pedram Razavi,Jorge Reis-Filho,Bob Li(图源:MSKCC)
我们都知道,随着人体各种细胞的死亡破裂,细胞的DNA会释放到血液中,这些DNA就构成循环游离DNA(cfDNA)。同理,肿瘤细胞也会死亡,并往血液中释放游离DNA,这就是我们所熟悉的循环肿瘤DNA(ctDNA)[3,4]。很显然,ctDNA只是cfDNA中带有基因变异的那一小部分[5-7]。
ctDNA的发现,让科学家对癌症的液体活检技术寄予厚望。
虽然科学家想到了很多方法研究ctDNA[8-10],但是仍难摆脱多种混杂因素的影响[11]。
其中影响比较大的一个是克隆性造血。
克隆性造血是那些有基因突变的造血干细胞,通过多系造血分化,形成携带同样突变的终末分化成熟血细胞[12]。虽然其中一些突变会导致后代血细胞具备适应性优势,出现不成比例的扩增[13],但是这种克隆性扩增并不是恶性的。
克隆性造血会随着年龄的增长而增加,之前有研究表明31%的老年人都会出现克隆性造血[14]。而且,导致克隆性造血的基因突变,也可以通过cfDNA检测到[15]。
Reis-Filho等认为,克隆性造血肯定会影响肿瘤学家对cfDNA测序结果的解释,尤其是很大一部分cfDNA片段源自造血细胞[16]。
▲ 图片来自pixabay.com
尽管大量的研究表明,通过cfDNA检测到的基因突变,与通过肿瘤组织检测到的基因突变有很好的一致性[10]。不过,还是有科学家在cfDNA中发现了很多肿瘤组织中不存在的基因突变[6]。当然啦,这部分突变有可能是因为组织活检取样的不全面带来的,也有可能是克隆性造血导致的。
关于这个问题,Reis-Filho等认为需要好好研究一番。
在2015年9月到2016年8月之间,研究人员一共招募了124名晚期未经治疗或者进行性转移的癌症患者,其中乳腺癌(MBC)患者39名,非小细胞肺癌(NSCLC)患者41名,前列腺癌(CRPC)患者44名。另外还招募了50名没有癌症的志愿者,作为对照样本,不过最终只有47名无癌志愿者的样本有评估价值。
研究人员从癌症患者身上获取了肿瘤组织样本、cfDNA和白细胞DNA,从无癌志愿者身上获取了cfDNA和白细胞DNA样本。
获取样本之后,他们基于已经获得FDA批准的MSK-IMPACT平台,给每个样本独立开展了深度高达60000×的测序(这个测序深度,成本该有多高……)。
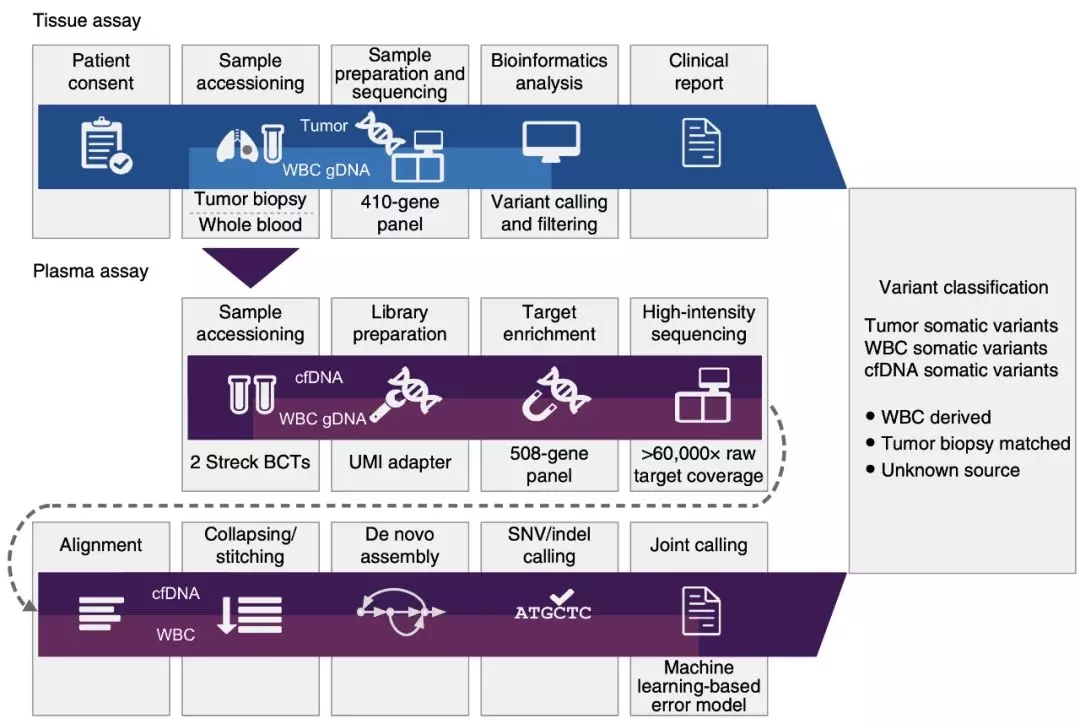
▲ 实验流程图
从总体上看,基于肿瘤组织的活检发现了740个突变,基于cfDNA的检测包含其中的530(71.6%)。具体到各个癌种的话,比例也是类似的,乳腺癌72%,非小细胞肺癌和前列腺癌是71%。
而且,在cfDNA发现的基因突变中,有一些突变虽然在肿瘤组织检测中出现,但是频率极低,还有一些在肿瘤组织和白细胞中均没有出现过(研究人员把它们叫做VUSOs)。
在确定了他们这个高深度测序方法的准确性和敏感性之后。研究人员推测,那些在肿瘤组织和白细胞中均没有出现过突变,可能反应了肿瘤的进化和异质性。而携带这部分变异的肿瘤组织,可能没有被活检采集到。
通过以上数据,我们不难发现,cfDNA检测和基于组织的活检还是存在一定的差异的。
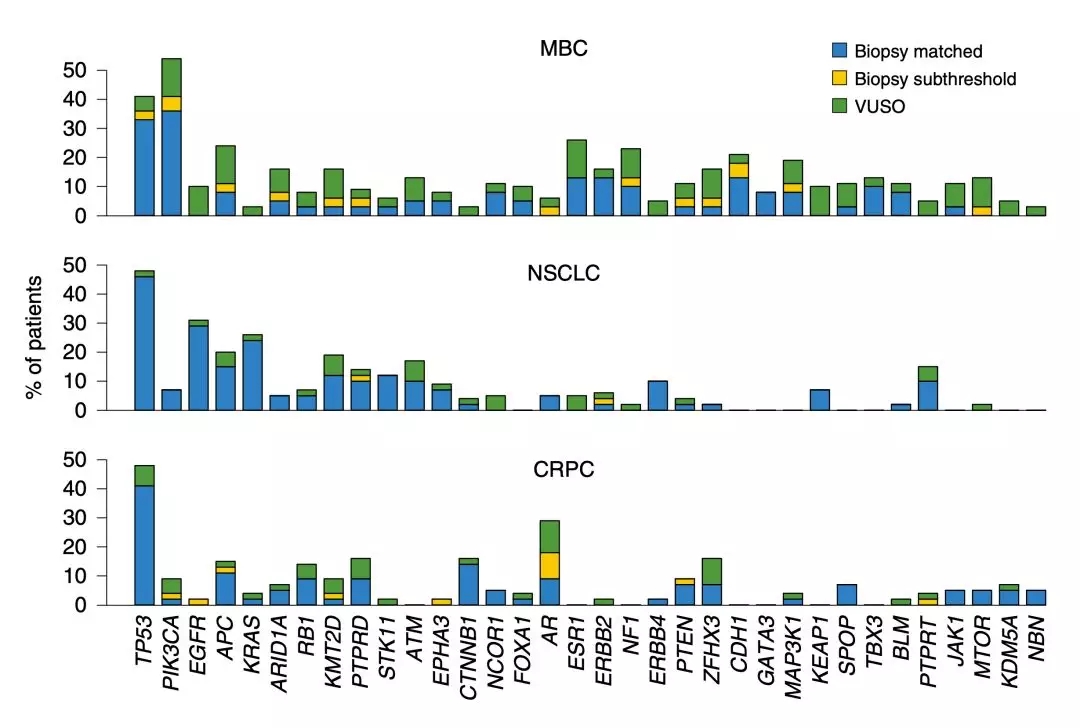
▲ cfDNA检测的主要基因突变分析
紧接着,研究人员又开展了一项探索性分析:是否可以通过cfDNA评估TMB、肿瘤突变特征和MSI。
分析发现,肿瘤组织活检发现了6个高TMB的样本,其中4例样本也被cfDNA判定为有高TMB。不过,cfDNA还判定另外6个样本为高TMB样本,但是肿瘤组织检测结果不支持cfDNA的结果。
在cfDNA确定的10个高TMB样本中,有75%的突变是在肿瘤组织中出现频率低,以及在肿瘤组织和白细胞中均没有出现过的。据此,研究人员进一步推测,这可能确实是肿瘤的空间异质性导致的,它们只在一些部位出现超突变,而这个部位恰恰没有被组织活检取到样。随后的基因分析也从侧面佐证了上述结果。
总结以上数据,研究人员认为,cfDNA测序可以准确检测整个肿瘤基因组中很大一部分的突变,或许可以反映患者肿瘤的TMB、肿瘤突变特征和MSI。而且,cfDNA可以检测到肿瘤组织活检发现不了的亚克隆突变。
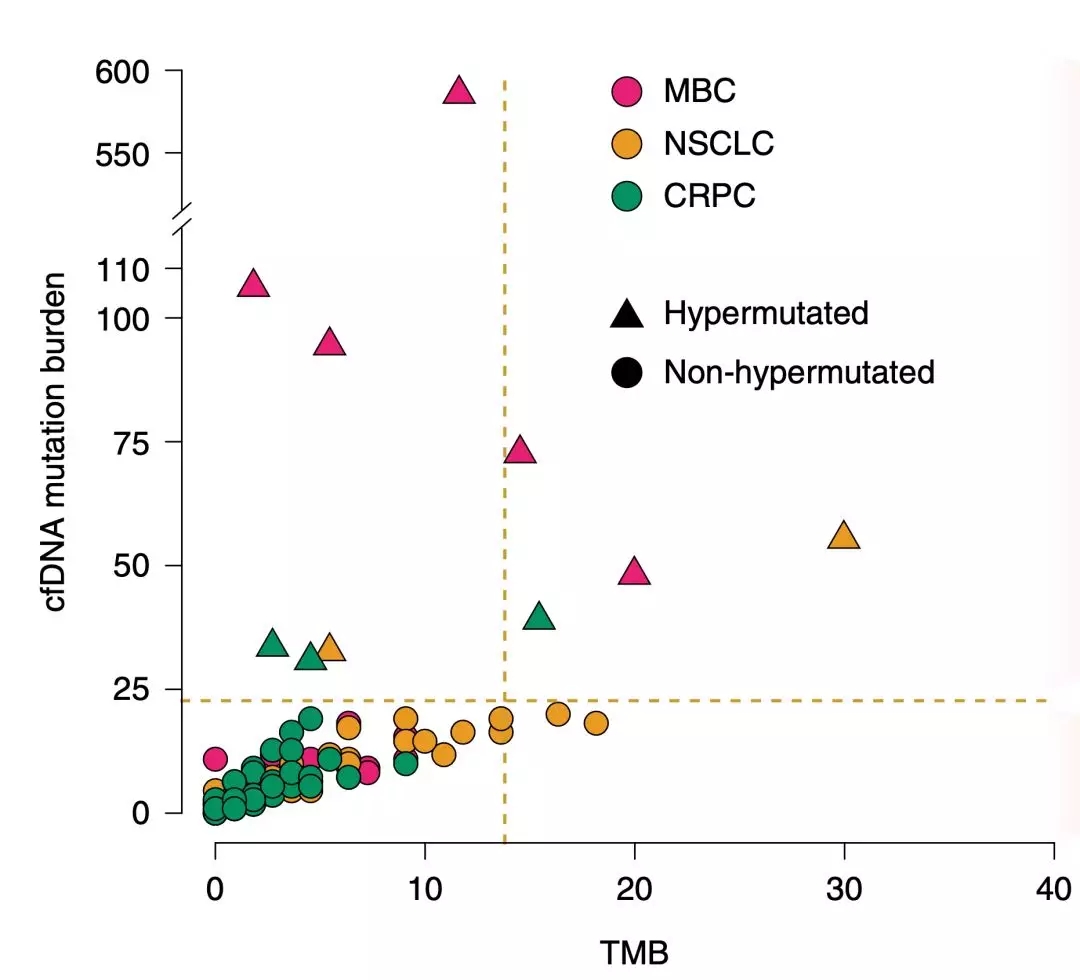
▲ TMB分布图
随后,研究人员又分析了cfDNA检测到的突变生物学来源。在无癌的志愿者样本中,cfDNA检测到的突变(364),有81.6%(297)出现在了白细胞DNA中;而在癌症患者群体中,cfDNA检测到的1727个突变,有53.2%(918)出现在了白细胞DNA中。
并且通过一系列的分析,研究人员认为,通过cfDNA进行超高深度测序发现的基因突变,不太可能是技术本身带来的假阳性。而且,之前研究中开展的的白细胞DNA低深度测序[14,17-20],可能大大低估了克隆性造血的普遍程度。
总之,在Reis-Filho团队看来,他们提出的这种超高深度cfDNA测序分析,确定克隆性造血突变是cfDNA中非肿瘤突变的最可能来源。而且这个比例相当惊人,至少在这个研究中,93.6%的无癌志愿者的白细胞携带一个克隆性造血突变,而这一比例在癌症患者群体中竟高达99.1%。
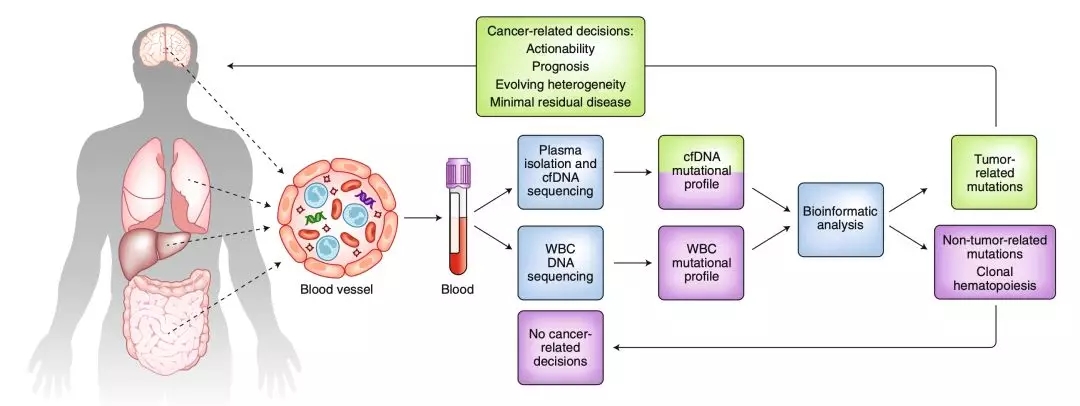
▲ 实验设计过程图
最后,论文作者在文章的讨论部分指出了一个有趣的现象。他们认为,当前大多数临床的cfDNA检测,都没有同时检测白细胞的DNA。结合着这个研究成果来看,这种不同时检测白细胞DNA的做法,肯定是不合适的。
不过,我今天特地了解了几个国内头部肿瘤测序公司的情况,基本都在检测cfDNA的同时,检测了白细胞的DNA,只是测序深度远低于MSKCC这个研究。
无论如何,这个研究一方面证明了cfDNA检测相较于组织活检的优点,另一方面也警示相关研究人员,在检测cfDNA的同时,一定要检测白细胞的DNA,最好是超高深度测序。
参考资料:
[1].https://www.mskcc.org/blog/many-mutations-detected-liquid-biopsy-tests-do-not-come-cancer-cells-msk-study-finds
[2].Razavi, P., Li, B.T., Brown, D.N, etal. High-intensity sequencing reveals the sources of plasma circulating cell-freeDNA variants[J]. Nature medicine, 2019. doi:10.1038/s41591-019-0652-7
[3].Leon S A, Shapiro B, Sklaroff D M, etal. Free DNA in the serum of cancer patients and the effect of therapy[J].Cancer research, 1977, 37(3): 646-650.
[4].Stroun M, Anker P, Lyautey J, et al.Isolation and characterization of DNA from the plasma of cancer patients[J].European Journal of Cancer and Clinical Oncology, 1987, 23(6): 707-712.
[5].Diaz Jr L A, Bardelli A. Liquidbiopsies: genotyping circulating tumor DNA[J]. Journal of clinical oncology,2014, 32(6): 579.
[6].Wan J C M, Massie C, Garcia-Corbacho J,et al. Liquid biopsies come of age: towards implementation of circulatingtumour DNA[J]. Nature Reviews Cancer, 2017, 17(4): 223.
[7].Adalsteinsson V A, Ha G, Freeman S S,et al. Scalable whole-exome sequencing of cell-free DNA reveals high concordancewith metastatic tumors[J]. Nature communications, 2017, 8(1): 1324.
[8].Jamal-Hanjani M, Wilson G A, McGranahanN, et al. Tracking the evolution of non–small-cell lung cancer[J]. New EnglandJournal of Medicine, 2017, 376(22): 2109-2121.
[9].Murtaza M, Dawson S J, Tsui D W Y, etal. Non-invasive analysis of acquired resistance to cancer therapy bysequencing of plasma DNA[J]. Nature, 2013, 497(7447): 108.
[10].Parikh A R, Leshchiner I, Elagina L,et al. Liquid versus tissue biopsy for detecting acquired resistance and tumorheterogeneity in gastrointestinal cancers[J]. Nature medicine, 2019, 25(9):1415-1421.
[11].Risques R A, Kennedy S R. Aging andthe rise of somatic cancer-associated mutations in normal tissues[J]. PLoSgenetics, 2018, 14(1): e1007108.
[12].Steensma D P, Bejar R, Jaiswal S, etal. Clonal hematopoiesis of indeterminate potential and its distinction frommyelodysplastic syndromes[J]. Blood, 2015, 126(1): 9-16.
[13].Bowman R L, Busque L, Levine R L.Clonal hematopoiesis and evolution to hematopoietic malignancies[J]. Cell stemcell, 2018, 22(2): 157-170.
[14].Jaiswal S, Fontanillas P, Flannick J,et al. Age-related clonal hematopoiesis associated with adverse outcomes[J].New England Journal of Medicine, 2014, 371(26): 2488-2498.
[15].Liu J, Chen X, Wang J, et al.Biological background of the genomic variations of cf-DNA in healthyindividuals[J]. Annals of Oncology, 2018, 30(3): 464-470.
[16].Hu Y, Ulrich B C, Supplee J, et al.False-positive plasma genotyping due to clonal hematopoiesis[J]. ClinicalCancer Research, 2018, 24(18): 4437-4443.
[17].Coombs C C, Zehir A, Devlin S M, etal. Therapy-related clonal hematopoiesis in patients with non-hematologiccancers is common and associated with adverse clinical outcomes[J]. Cell StemCell, 2017, 21(3): 374-382. e4.
[18].Jaiswal S, Natarajan P, Silver A J, etal. Clonal hematopoiesis and risk of atherosclerotic cardiovascular disease[J].New England Journal of Medicine, 2017, 377(2): 111-121.
[19].Xie M, Lu C, Wang J, et al.Age-related mutations associated with clonal hematopoietic expansion andmalignancies[J]. Nature medicine, 2014, 20(12): 1472.
[20].Genovese G, Kähler A K, Handsaker R E,et al. Clonal hematopoiesis and blood-cancer risk inferred from blood DNAsequence[J]. New England Journal of Medicine, 2014, 371(26): 2477-2487.
(来源于: 奇点糕 奇点网)


